王久存课题组合作研究揭示中国汉族人群皮肤微生物组特征
人类遗传学与人类学系王久存教授课题组和深圳华大生命科学研究院刘晓课题组合作研究揭示中国汉族人群皮肤微生态宏基因组特征,为进一步探索皮肤微生物功能、利用皮肤微生物改善皮肤状态提供了理论基础。2021年2月17日,相关研究成果以“Characterization of the human skin resistome and identification of two microbiota cutotypes”为题在线发表于Microbiome杂志。

主要亮点如下:①采用宏基因组测序法检测了822个健康汉族人群面部皮肤菌群样本,结合HMP数据,构建了首个皮肤微生物基因集iHSMGC;②表皮葡萄球菌属是皮肤菌群耐药基因的重要来源,耐药基因的分布具有微生物特异性和部位特异性;③发现了中国汉族人群显著富集的一种皮肤微生物:奥斯陆莫拉菌;④基于皮肤菌群鉴定出了2种广泛存在的皮肤亚型:C型和M型,分别以痤疮丙酸杆菌和奥斯陆莫拉菌为指示菌;⑤两种皮肤亚型表现出不同代谢功能特征,可能与宿主提供的营养不同有关。
皮肤被认为是一个复杂的“生态系统”,栖息着各种各样的细菌、真菌和病毒。这些微生物的集合被称为皮肤微生物组或皮肤菌群(skin microbiota),对于维持宿主皮肤稳态和正常皮肤屏障功能有重要作用,菌群失调可引起一系列皮肤疾病。此前的皮肤菌群研究主要集中在欧美高加索人群,针对亚洲人群的皮肤菌群研究仍较少,尤其缺乏宏基因组学研究。
在Microbiome的这项最新研究中,研究团队首次对中国汉族人群的皮肤菌群进行了大样本研究。对来自约300个健康个体的面部(额头、鼻旁、面颊)共计822个样本进行宏基因组测序,结合来自HMP公开数据的15名北美健康个体20余个部位共计538个宏基因组测序样本,建立了首个跨人群的人类皮肤微生物组基因集(integrated Human Skin Microbial Gene Catalog,iHSMGC).
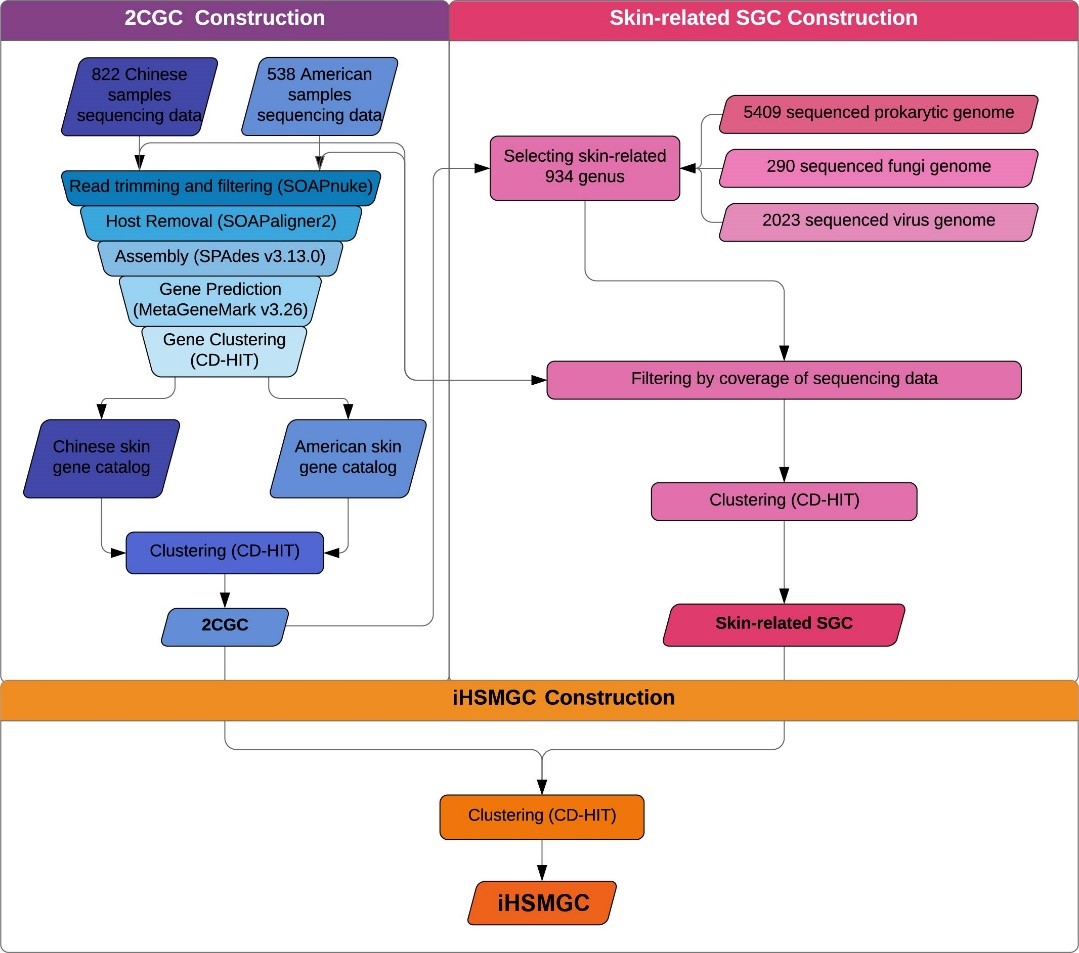
图1. 人皮肤微生物组基因集(iHSMGC)构建流程图
iHSMGC包含10,930,638个基因,相比于北美人群,复旦及华大团队鉴定出4,879,024个新基因。基因集不仅为后期皮肤宏基因组研究的数据分析提供了重要工具,也极大拓宽了我们对皮肤微生物功能多样性的认知。有趣的是基因丰度最高的基因富集于辅助因子和维生素的生物合成,该功能不仅对微生物的生长和生存至关重要,维生素的合成亦与皮肤健康紧密相关。此外,基因集中可见依克多因(Ectoin)等已应用于美白抗衰的生物活性分子,提示皮肤微生物组基因集是一个有待开发的巨大宝藏。
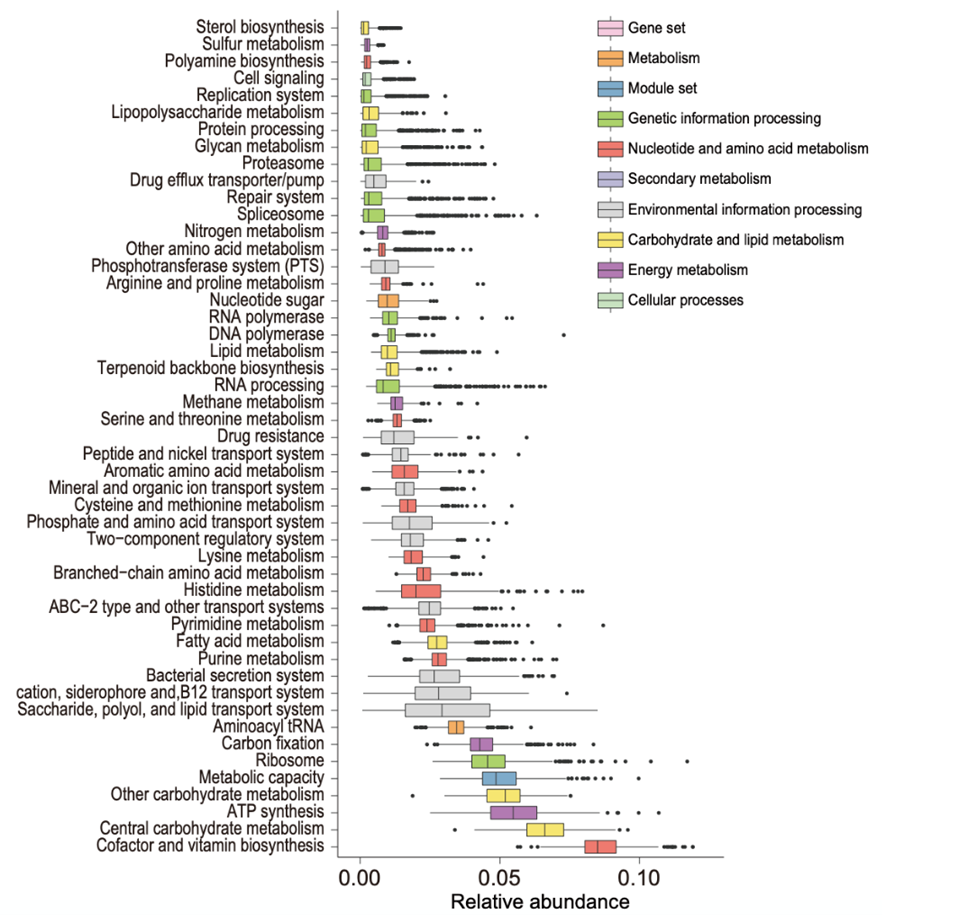
图2. 中国人群皮肤微生物组基因的功能富集
抗生素的滥用导致耐药细菌的出现和广泛传播,每年近百万人死于耐药菌造成的感染性疾病,并给全世界经济造成极大损失。面对越来越严峻的细菌耐药性形势,加强耐药菌株的监控以及耐药菌株特征的分析显得尤为重要。精确地获得耐药菌株的DNA信息(包括耐药基因、毒力因子等特征)对于耐药菌株的分子流行病学和传播机制研究至关重要。传统的技术手段如PCR和脉冲场凝胶电泳(Pulsed field gel electrophoresis,PFGE)等得到的结果不够全面且精确度低。宏基因组测序技术结合生物信息学分析成为研究细菌耐药性的重要技术手段。该项研究首次展示了健康中国人群皮肤微生物组的耐药抗性组(human skin resistome),发现正常皮肤共生菌如多种葡萄球菌(Staphylococcus spp)是体内重要的耐药基因仓库。整体而言,中国人群的耐药基因(Antibiotic resistance genes,ARGs)数量显著高于北美人群,但在两种人群中均发现极低丰度的多重耐药超级细菌。有趣的是,耐药基因有显著的部位差异性。
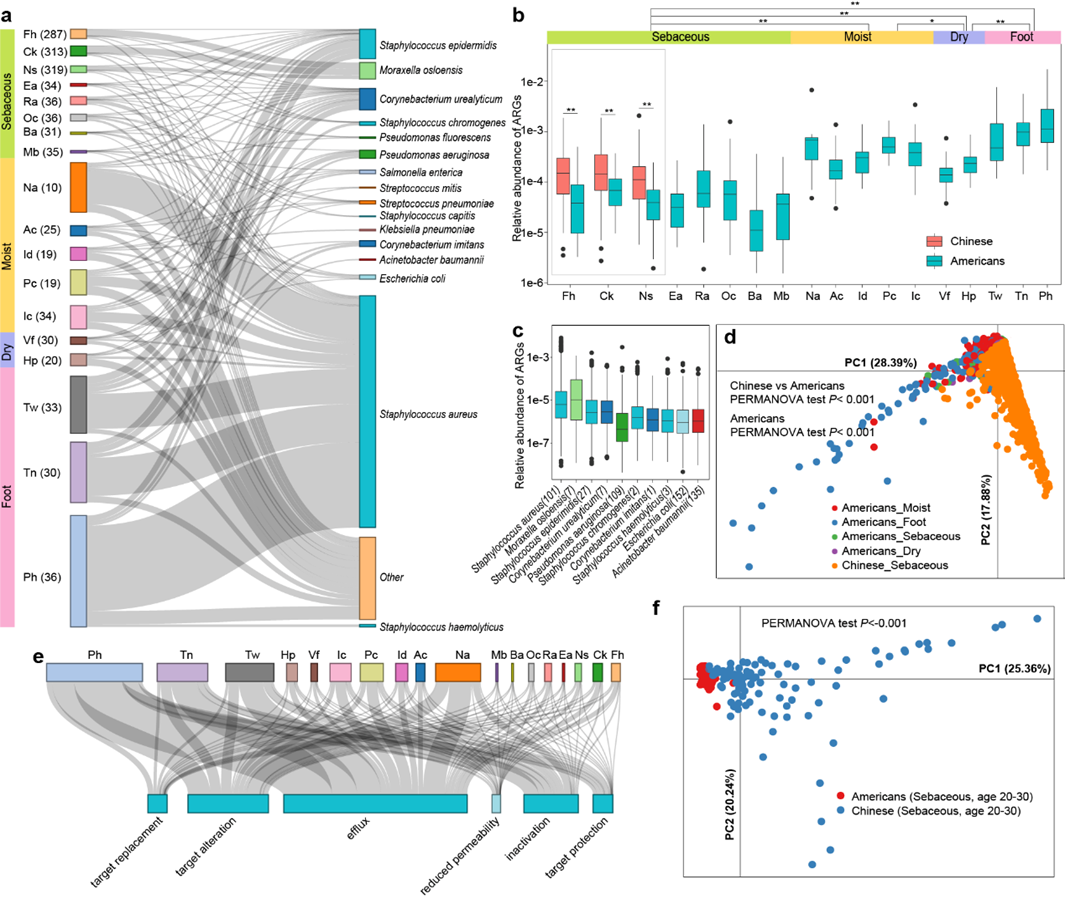
图3. 皮肤菌群的耐药基因
与北美人群的皮肤菌群类似,中国人群中痤疮丙酸杆菌、表皮葡萄球菌、马拉色菌属、痤疮丙酸杆菌噬菌体等是主要的皮肤微生物;皮肤微生物间存在着显著的个体差异性;对照北美数据,研究者发现7 种细菌普遍存在于所有样本中,与部位及种族无关:拟棒状杆菌、痤疮丙酸杆菌、颗粒表皮杆菌、金黄色葡萄球菌、头状葡萄球菌、表皮葡萄球菌、肺炎链球菌,提示这些菌可能在皮肤上起到相对保守的作用。此外,本项研究首次发现了一种在中国人群中明显富集的微生物物种:奥斯陆莫拉菌(Moraxella osloensis)。
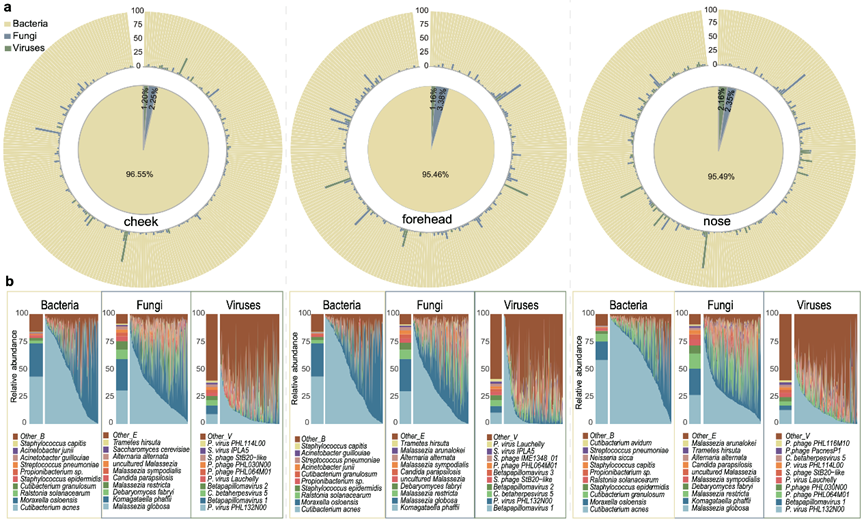
图4. 中国汉族人群面部皮肤微生物组成
此外,研究团队发现两类人群的皮肤微生物组均存在极强的个体差异性。面对如此复杂的多样性,若能解析微生物群落组成的结构模式则可大大降低数据的复杂性。群体分型(stratification)已应用于肠道微生物中,被定义为“肠型”(enterotypes),可有效描述肠道微生物群落结构,对指导临床实践产生了积极的影响(Nature Microbiology 2017)。在本项研究中,研究者首次报道皮肤微生物在中国人群的群体水平亦存在2种稳定的群落组成模式,将其命名为“皮肤型”(cutotypes),分别是以奥斯陆莫拉菌(Moraxella osloensis)为主导的M型皮肤(M-cutotype)和以痤疮丙酸杆菌(Cutibacterium acnes)为主导的C型皮肤(C-cutotype)。
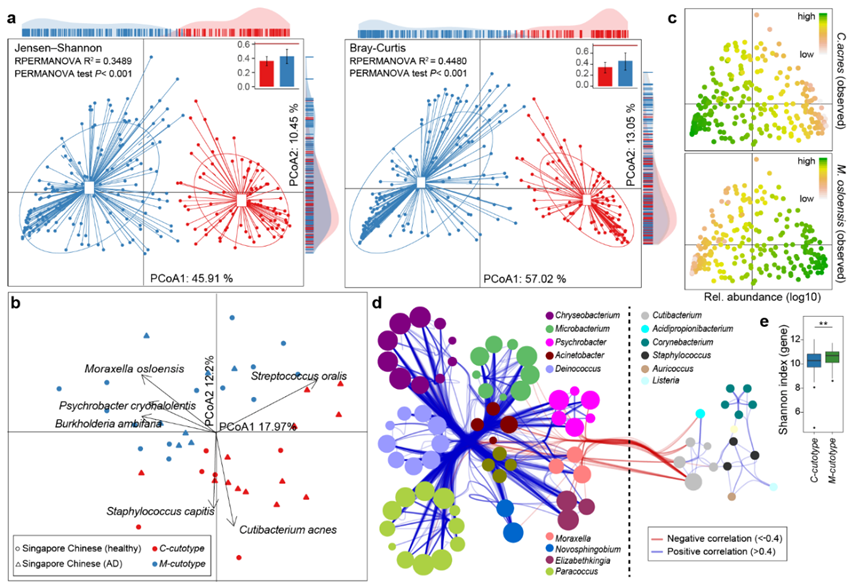
图5. 中国汉族人群基于皮肤菌群的皮肤分型(cutotypes)
两种皮肤型具有不同的微生物网络特征,皮肤型内部的微生物之间呈正相关,与外部微生物呈负相关。宏基因组基因功能富集提示C型皮肤微生物群落富集碳水化合物和甾醇代谢、脂肪酸合成等,M型皮肤微生物富集氨基酸、芳香族化合物代谢、耐药性等。进一步的宿主临床表现也显示出明显差异:C型皮肤水分油脂含量高,年轻人居多;而M型皮肤水分油脂含量偏低,肤色暗沉偏黄,老年人居多。基于皮肤型的这一新式分型将大大简化微生物个体差异性的评估,方便人们抓住核心模块的主要差异特征,进而实现潜在的个性化诊断和精准干预。

图6. 皮肤分型的基因功能富集差异及宿主皮肤特征差异
华大健康研究院李志明、王久存课题组博士后夏晶晶、硕士研究生蒋刘一琦及博士研究生谈益妹为本文共同第一作者,复旦生科院教授王久存、生科院讲座教授及德国莱布尼兹环境医学研究所院士Jean Krutmann、华大生命科学研究院研究员刘晓(现清华大学深圳国际研究生院副教授)和聂超为本文的共同通讯作者。本研究获得了上海市级重大专项、国家自然科学基金、国家重点研发计划、中国医学科学院创新单元等项目的资助。
原文链接:https://link.springer.com/content/pdf/10.1186/s40168-020-00995-7.pdf
文章简介视频链接:https://vimeo.com/503648046
关键词:宏基因组、皮肤微生物组、基因集、抗性组、抗性基因、奥斯陆莫拉菌、皮肤型
Keywords: shotgun metagenomic sequencing; skin microbiome; gene catalog; resistome; antibiotic resistance genes (ARGs); Moraxella osloensis; cutotypes
