《自然·生物技术》刊发国际组学数据质量控制联盟最新研究成果

美国东部时间2021年9月9日上午11:00,国际权威学术期刊《自然·生物技术》(Nature Biotechnology)在线发表了5篇最新科研论文,专题发布了国际组学数据质量控制联盟第四期(MAQC-IV,亦为“测序质量控制第二期SEQC2”)的最新研究成果。
该项目由美国食品药品监督管理局(FDA)牵头,历时5年多,聚集了来自全球100多个单位的300多位科学家参与。通过建立和使用一系列基因组参考物质,国际组学数据质量控制联盟对众多新一代测序平台和生物信息学方法在体细胞和胚系突变、拷贝数变异、肿瘤靶向测序和液态活检、单细胞测序等多个应用场景的性能进行了系统研究。人类遗传学与人类学系石乐明教授团队是国际组学数据质量控制联盟项目的核心推动者之一。在本次《自然·生物技术》杂志刊发的5篇论文中,石乐明教授本人作为共同通讯作者,参与领导了其中2篇论文的研究与撰写工作。
长久以来,由于缺乏合适的参考物质,无法产生标准化的DNA测序数据集并对不同测序流程或不同算法的性能进行全面测试和评估,因而制约了肿瘤基因组学方法的落地实施和广泛使用。在建立共享的参考样本、数据和变异集合以便使用全基因组测序对肿瘤突变检测进行全面评估(Establishing community reference samples, data and call sets for benchmarking cancer mutation detection using whole-genome sequencing)的论文中,中外科学家聚焦了这一科研难点。
首先,科研团队基于一株高度异质性并富含体细胞突变的三阴性乳腺癌细胞系及其配对的正常淋巴母细胞系,建立了基因组DNA参考物质;随后利用多个全基因组测序平台、多种建库方法和多种生物信息学手段获得了全基因组范围内的高置信体细胞突变和胚系变异,并利用不同全外显子组测序平台和高深度靶向测序进行了验证。尽管来自细胞系的基因组DNA参考物质不能完全代表临床样本中的原发肿瘤细胞,但在建立测序流程并对其可靠性进行客观评估时,不仅可以最大限度地减少来自技术、分析和信息学的潜在偏倚,还可以为“仅用肿瘤”或“匹配肿瘤-正常样本”进行体细胞突变分析的方法验证提供独特的资源。
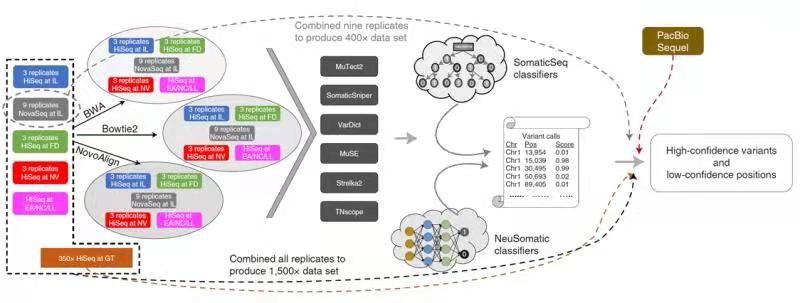
图1 Schematic of the bioinformatics pipelines used to define the confidence levels of the somatic mutation call set
罗氏公司的Li Tai Fang、美国国家癌症研究所(NCI)的Bin Zhu和Yongmei Zhao为该文共同第一作者;美国食品药品监督管理局的Wenming Xiao和Huixiao Hong、洛马林达大学的Charles Wang以及人类遗传学与人类学系的石乐明为该文共同通讯作者。
在另一篇题为利用全基因组和全外显子组测序进行癌症突变检测的最佳实践(Toward best practice in cancer mutation detection with whole-genome and whole-exome sequencing)的论文中,科学家们则聚焦如何开发精准肿瘤医学临床应用的可靠检测方法,从而真正发现有意义的肿瘤特异性突变而非新一代测序流程中各环节可能引发的技术误差。
此前,生物医学界并没有大规模的测序研究涉及跨实验室重复性的问题,也未曾系统地探讨生物学、技术和计算因素是如何影响对体细胞突变的识别。基于上一篇论文研究中建立的配对肿瘤-正常细胞系基因组DNA参考物质,中外科学家在本篇论文中对影响体细胞突变检测在6个不同测序中心的重复性和准确性的众多因素进行了系统研究。科学家们使用全基因组测序和全外显子组测序,评估了不同样本类型、不同样本用量、不同肿瘤纯度、不同文库构建方案、不同生物信息学分析方法对突变结果的影响。发现全基因组测序和全外显子组测序结果的可重复性均受测序深度和突变检出方法的影响,但全外显子组测序的性能还受插入片段大小、基因组拷贝数和基因组总体不平衡评分 (GIV, G>T/C>A)的影响。最后,通过同时考虑文库制备方法、肿瘤含量、测序深度和生物信息学方法对突变结果的影响,推荐了可操作的实践指南,以提高新一代测序在肿瘤突变检测中的可重复性和准确性。
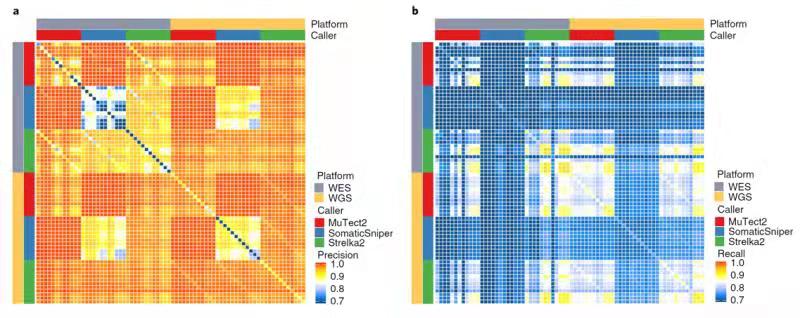
图2 Biological repeats versus analytical repeats
美国食品药品监督管理局的Wenming Xiao和复旦大学人类表型组研究院的任路瑶为该文共同第一作者,石乐明、洛马林达大学的Charles Wang、美国食品药品监督管理局的Huixiao Hong和Wenming Xiao为该文共同通讯作者。
在刊发上述系列成果和论文的同时,《自然·生物科学》还同期配发了主编Andrew Marshall撰写的社论,认为测序质量控制二期研究(SEQC2)对新一代测序平台和分析方法进行了迄今为止最为全面客观的评估,为基础研究、临床和监管部门阐明了相关的技术规范和质量控制指标;自2005年启动以来,国际组学数据质量控制联盟根本上改变了基因组数据分析的实践,是共享利他和开放科学的绝妙例子,应成为其它快速发展的技术领域(如宏基因组学、空间转录组学和蛋白质组学等)努力学习的样板。
同一期杂志还刊发了由斯坦福大学教授Marc Salit(原美国国家标准与技术研究院GIAB瓶中基因组项目负责人)和美国食品药品监督管理局执行局长Janet Woodcock合作撰写的评论——“国际组学数据质量控制联盟与基因组学医学时代(MAQC and the era of genomic medicine)”。该文认为,国际组学数据质量控制联盟为期16年的组学数据质量控制国际合作科研项目,提升了人们对基因组数据可靠性的信心,直接促进了监管科学的发展,证明监管科学可以促进新技术的快速推广和临床使用,正支撑着精准医学领域的许多药物和诊断设备的上市申报。该文还指出,国际组学数据质量控制联盟是促进技术成功转化的一个重大案例,希望其成功将在更多研究领域得以复制,克服从技术到产品转化过程中的障碍。
国际组学数据质量控制联盟(MicroArray Quality Control Consortium),简称MAQC,由时任美国食品药品监督管理局高级研究员石乐明教授于2005年发起成立,其全职进入复旦大学担任教授后,继续积极推动联盟的发展和全球生物医学界在组学数据质量控制上的大力合作。该联盟旨在通过质量控制和标准化手段,确保基因芯片数据产生、分析和解读等诸多环节的可靠性,随后扩展到新一代测序质量控制(SEQC, SEquencing Quality Control)。至今为止,该联盟就基因表达谱数据质量控制(MAQC-I)、生物标志物和预测模型挖掘的最佳实践(MAQC-II)、转录组测序数据质量控制(MAQC-III/SEQC1)以及基因组DNA测序数据质量控制(MAQC-IV/SEQC2)进行了长期而系统的探索,其研究成果由《自然·生物技术》杂志分别于2006年、2010年、2014年和2021年以4个专辑发表,促进了FDA基因组学指南和有关国际标准的制定。
在此基础上,国际组学大数据质量控制学会 (The International Massive Analysis and Quality Control Society)于2017年4月12日成立(www.maqcsociety.org;Shi L et al., Nature Biotechnology 2017),并于2018年2月在复旦大学成功举办了全球学术年会。该学会旨在通过建立共享可靠的多组学(包括基因组学、转录组学、蛋白质组学、代谢组学等)参考物质和与研究目标密切相关的质量评价指标,确保高通量组学技术的可重复性和可靠性,为精准医学保驾护航。16年来,我国多家高校、科研院所、企业和医院的数十名科研工作者积极参与了国际组学数据质量控制联盟的工作,在提升我国组学数据质量意识、引领国际标准研发、推动我国生物医学领域原创研究的过程中起到了重要的作用。

图3 国际MAQC组学大数据质量控制学会第二届学术研讨会
相关论文原文链接:
1. Establishing community reference samples, data and call sets for benchmarking cancer mutation detection using whole-genome sequencing
https://www.nature.com/articles/s41587-021-00993-6
2. Toward best practice in cancer mutation detection with whole-genome and whole-exome sequencing
https://www.nature.com/articles/s41587-021-00994-5
![]() 斯坦福大学教授Marc Salit和FDA执行局长Janet Woodcock发表评论
斯坦福大学教授Marc Salit和FDA执行局长Janet Woodcock发表评论
