倪挺团队开发新算法揭示基因内含子多聚腺苷酸化的动态调控
基因组领域知名期刊《Genome Research》在2021年11月刊发表了复旦大学倪挺课题组题为“Cancer-associated dynamics and potential regulators of intronic polyadenylation revealed by IPAFinder using standard RNA-seq data”的方法学研究论文。该研究开发了可以基于常规RNA-seq数据推断基因中内含子poly(A)位点并定量其使用的生物信息学方法IPAFinder,利用IPAFinder可以帮助探索内含子多聚腺苷酸化在不同病理和生理条件下的动态变化和潜在生物学功能。

内含子多聚腺苷酸化(Intronic polyadenylation,IPA)指基因使用内含子poly(A)位点从而产生截短转录本的现象。最近的研究表明IPA在白血病发生发展和肌肉纤维化过程中具有重要作用。基于常规RNA-seq数据推断位于3′ UTR区域的可变多聚腺苷酸化的方法(比如DaPars和QAPA)已有开发,但仍没有基于使用最广泛的常规RNA-seq数据推断内含子中多聚腺苷酸化的方法,阻碍了对基因非编码区复杂调控的认识。
为了解决这一问题,本研究开发了IPAFinder的方法,可从常规RNA-seq数据中直接推断内含子poly(A)位点,并且不需要任何poly(A)位点的注释信息。模拟数据的测试证明IPAFinder不仅可以准确推断两种不同类型的IPA(composite IPA和skipped IPA),还可以排除内含子保留、可变5′剪切位点等选择性剪切事件的干扰。对于覆盖度在40×的有重复的模拟数据,IPAFinder能以高的准确率推断出超过80%的差异使用的IPA位点。通过比较分析同时拥有常规RNA-seq和3′-seq的真实数据,发现IPAFinder分析结果和3′-seq分析结果总体一致。此外IPAFinder还能检测到3′-seq检测不到的动态变化的IPA事件(比如基因PDXDC1的IPA事件)。
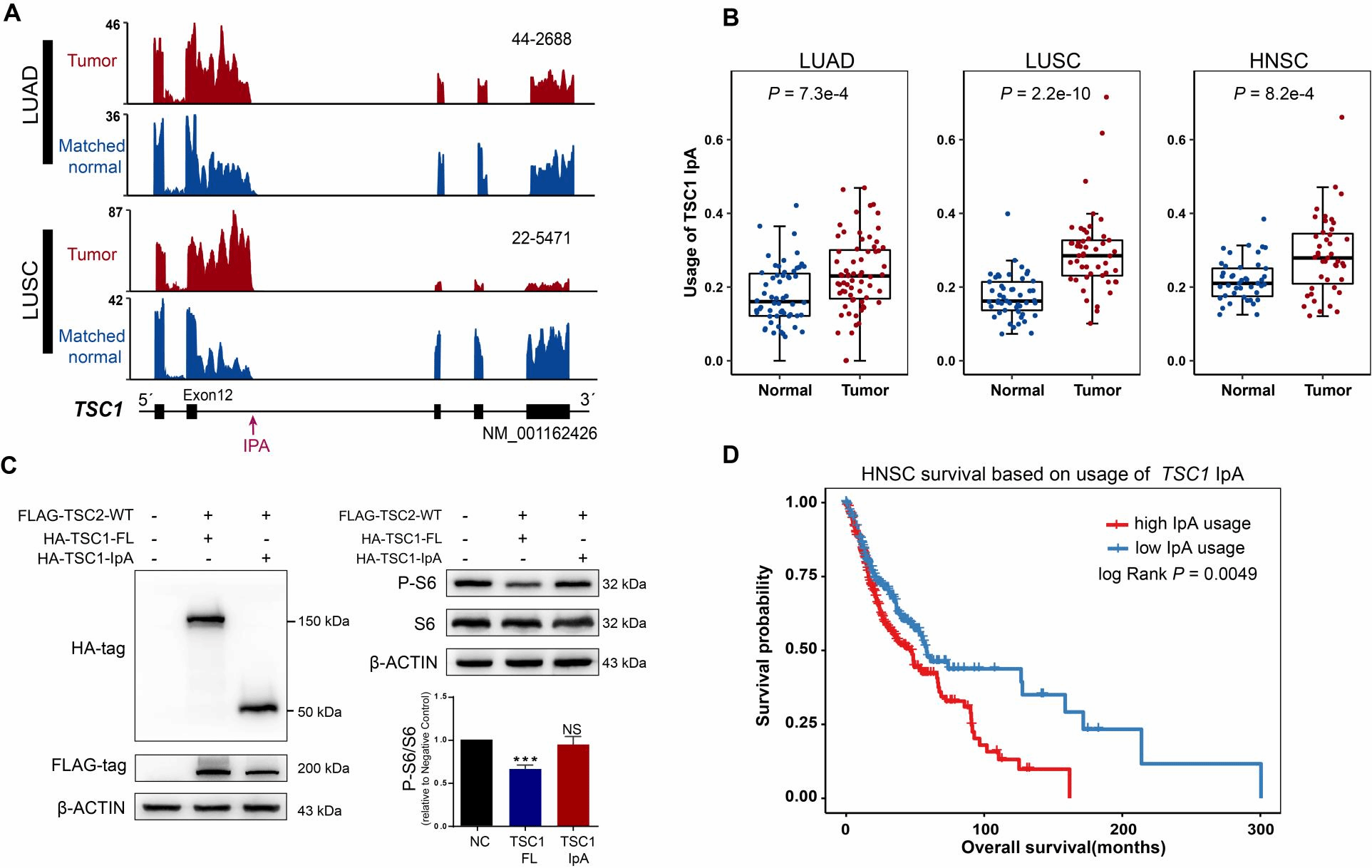
抑癌基因TSC1存在内含子多聚腺苷酸化的动态调控
通过使用IPAFinder分析TCGA数据库中六种癌症类型的正常/肿瘤配对样本RNA-seq数据,研究团队发现肿瘤样本中内含子poly(A)位点使用比例存在上调趋势。通过筛选,研究团队最终得到490个非冗余的动态变化的IPA事件,其中癌症相关基因TSC1、SPRED2和CCND2均被发现存在新的IPA调控。研究团队进一步通过湿实验分别对TSC1和SPRED2的IPA转录本的功能进行了探究,发现TSC1的IPA转录本可产生截断蛋白,但该截断蛋白与TSC1全长蛋白相比失去了抑制S6磷酸化的能力,这意味着TSC1的IPA转录本产生的截断蛋白不能像TSC1全长蛋白一样抑制mTOR信号通路。算法预测和实验验证均表明SPRED2的IPA转录本会产生非编码RNA,它与SPRED2全长转录本相比也失去了抑制肺癌细胞增殖的能力。此外,通过分析公共数据库,作者发现与细胞衰老相关的剪接因子(PTBP1/2、HNRNPC、U2AF1和U2AF2)以及与m6A修饰相关的因子(YTHDC1和METTL3)会调控内含子poly(A)位点的使用,提示IPA存在于众多的生物学过程中并可能发挥重要作用。
倪挺课题组博士生赵昭昭负责本研究的算法开发及生信分析部分,博士生许秋实负责实验验证部分,倪挺教授和生命科学学院魏刚博士为论文共同通讯作者。上海科技大学张力烨教授和复旦大学胡跃清教授为该研究提供了宝贵建议。该研究获得国家重点研发计划课题、国家自然科学基金项目及上海市首批市级科技重大专项“国际人类表型组计划(一期)”的支持。

论文链接:https://genome.cshlp.org/content/31/11/2095.long
